
-

生物通官微
陪你抓住生命科技
跳动的脉搏
Systematic Biology | 张蔚课题组和合作者开发基于深度学习的基因渐渗推断...
【字体: 大 中 小 】 时间:2023年06月07日 来源:生命科学联合中心
编辑推荐:
? Systematic Biology | 张蔚课题组和合作者开发基于深度学习的基因渐渗推断方法 解决分类群之间的演化关系是演化生物学的基本任务之一。在大数据时代,随着测序技术和分析方法的发展,多项研究发现类群之间的杂交和基因渐渗可能强烈的影响了生命之树,并在物种形成和适应过程中起到了重要...
解决分类群之间的演化关系是演化生物学的基本任务之一。在大数据时代,随着测序技术和分析方法的发展,多项研究发现类群之间的杂交和基因渐渗可能强烈的影响了生命之树,并在物种形成和适应过程中起到了重要作用。然而,现有基因渐渗检测方法仍具有一定局限性,如依赖大样本量、受限的系统发育关系或精确的种群演化历史等。因此,在基因流广泛存在的背景下,评估物种之间的复杂关系仍具挑战。同时,基于深度学习的算法已经迅速成为处理统计应用的有效替代方法,特别是与大数据集有关的应用。目前已有涉及基因渐渗推断的基于深度学习的相关方法的应用报道,但多是针对具体分类群的初步尝试,其一般适用性和可推广性受到了限制。
2023年6月1日,北京大学生命科学学院、蛋白质与植物基因研究国家重点实验室、北大-清华生命科学联合中心张蔚课题组,在Systematic Biology杂志在线发表了题为“Inferring historical introgression with deep learning”的研究论文,提出了一种基于深度学习算法检测基因渐渗的方法ERICA。该方法基于基因组序列信息,通过识别拓扑结构不一致鉴定基因组内的渐渗区域。该方法能够有效的在模拟数据和多种动植物分类群的真实基因组数据中检测渐渗信号,为利用基因组数据评估类群之间的演化关系提供了一种具有一般适用性的新方法,有助于推动杂交和基因渐渗相关的演化研究。
研究构建了处理序列比对数据的卷积神经网络模型,以定量评估类群之间的演化关系,并通过识别特定基因流对应的拓扑结构推断渐渗区域(图1)。研究首先利用模拟数据集对神经网络进行了训练和评估,并与其他广泛使用的基因渐渗检测方法进行了对比。评估结果显示神经网络模型能够有效地检测渐渗特别是适应性渐渗信号,且具有较好的泛化能力(图2)。
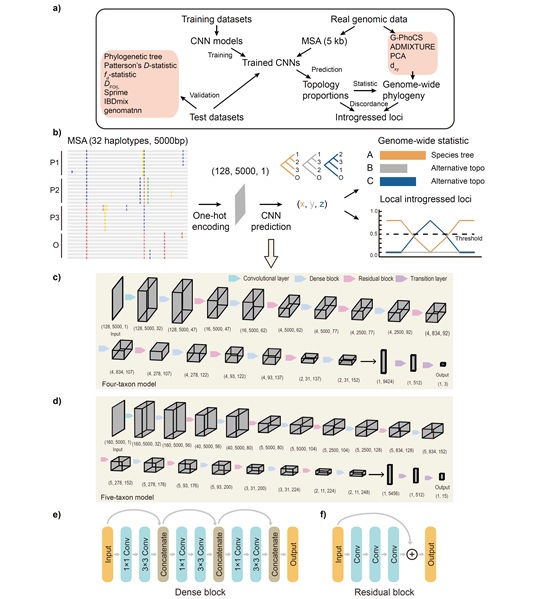
图1 ERICA方法设计原则和神经网络结构

图2 ERICA方法准确率评估
此外,研究者使用ERICA方法分析了适应性辐射类群袖蝶属(Heliconius)的群体基因组数据,并鉴定获得了已知的翅图案模式相关渐渗位点,表明ERICA方法能够在真实数据中检测到适应性渐渗信号,且具有较传统方法更小的随机误差。
之后,研究者展示了ERICA方法的一个应用实例,通过将该方法应用于稻属(Oryza)基因组比对及泛基因组数据分析,获得了粳稻和籼稻之间以及从野生稻(O. rufipogon)到籼稻的渐渗位点,其中包括多个与水稻驯化性状相关的受选择区域,支持基因流在籼稻驯化过程中起到重要作用(图3)。此外,研究发现籼稻和野生稻(O. nivara)与热带粳稻之间存在基因流,且渐渗基因可能与热带粳稻对热带环境的适应与抗逆性有关。

图3 稻属物种基因组渐渗模式
综上所述,该研究开发了一种新的基因渐渗检测方法,其能够高效处理基因组序列数据,准确推断局部的渐渗信号,有助于深入理解类群之间的复杂演化历史以及基因渐渗在物种适应中发挥的作用。值得一提的是,为了满足研究人员的广泛要求,该方法包括了一个在线提交门户和一个本地版本的工具包。因此,该研究不仅提供了一种先进的基因渐渗的研究方法,并且给出了完整的研究解决方案,展示了其解决多个系统中实际问题的能力。
北京大学生命科学学院/北大-清华生命科学联合中心张蔚研究员和北京脑科学与类脑研究中心张力研究员为本文共同通讯作者;北大-清华生命科学联合中心博士生张宇博、北京脑科学与类脑研究中心工程师朱庆杰为本文共同第一作者;北京脑科学与类脑研究中心工程师邵毅、北大-清华生命科学联合中心博士生姜焱晨和华中农业大学生命科学技术学院欧阳亦聃教授对本研究做出了重要贡献。该项目得到国家自然科学基金、北京市自然科学基金、北大-清华生命科学联合中心、蛋白质与植物基因研究国家重点实验室等资助。
原文链接:https://doi.org/10.1093/sysbio/syad033