
-

生物通官微
陪你抓住生命科技
跳动的脉搏
综述:VarMeter:一种预测糖基因变异影响的方法
【字体: 大 中 小 】 时间:2025年07月09日 来源:Journal of Human Genetics 2.6
编辑推荐:
这篇综述系统介绍了VarMeter系列工具如何通过整合蛋白质三维结构参数(如突变能量△△G、溶剂可及表面积nSASA、AlphaFold的pLDDT置信度评分)和Mahalanobis距离分析,提升糖基因(glycogene)错义变异致病性预测的准确性。文章重点阐述了该框架在先天性糖基化障碍(CDG)等疾病中的应用价值,为解析糖基化相关蛋白(如ARSL、SGSH、FUT3、POMK)的基因型-表型关系提供了新思路。
糖链作为结构最多样的生物分子,在细胞通讯、免疫应答等生理过程中发挥关键作用。糖基因(glycogene)的缺陷会导致先天性糖基化障碍(CDG),但约50%的糖基因功能尚未明确,部分归因于人类特有的糖基化特征难以通过模式生物完全模拟。随着AlphaFold等蛋白质结构预测技术的突破,基于结构的错义变异分析成为破解这一难题的新途径。
蛋白质稳定性可通过吉布斯自由能(△G)量化,错义变异通过改变△△G(△G突变体-△G野生型)破坏折叠平衡。如图1所示,△△G>0表示变异 destabilizes 蛋白结构。FoldX等工具能计算△△G,但误差达±0.85 kcal/mol,且受实验条件异质性限制。
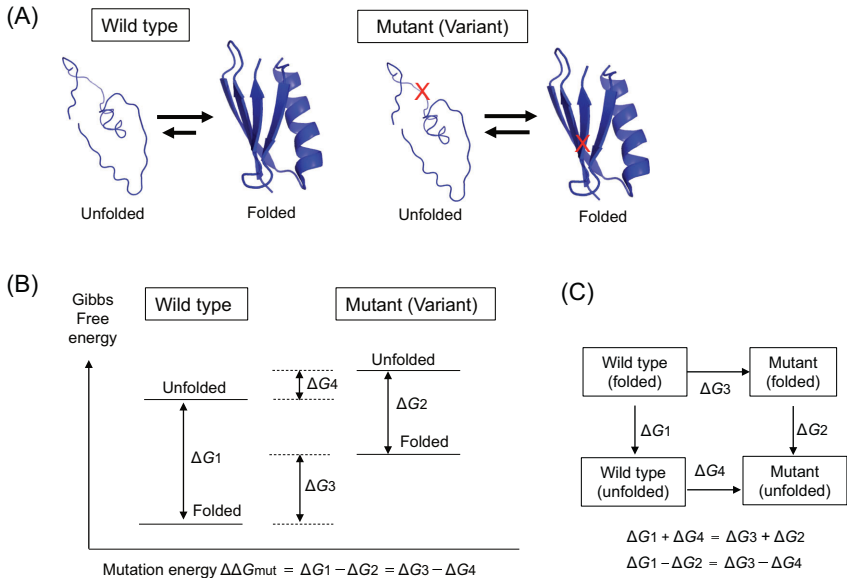
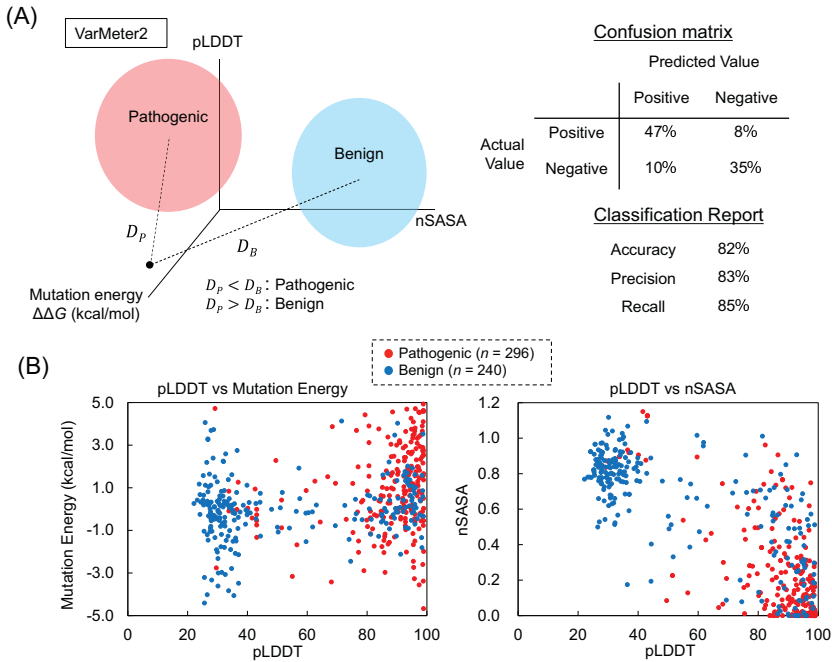
VarMeter创新性地引入归一化溶剂可及表面积(nSASA),通过"滚动水球"算法(图2)量化残基埋藏程度。当△△G≥0.88 kcal/mol且nSASA≤0.11时,变异被判定为致病性。在ClinVar数据集测试中,其准确率达74%,显著优于单一参数分析。

升级版VarMeter2新增pLDDT评分(反映局部结构置信度)和Mahalanobis距离分析,通过三维参数空间聚类(图4)将准确率提升至82%。例如对Sanfilippo综合征相关SGSH基因的Q365P变异,其预测的致病性(△△G=7.2 kcal/mol,nSASA=0.03)与实验验证的酶活丧失完全吻合。
在ARSL(导致CPDX1)案例中,VarMeter准确识别R111H等致病变异(图5),其预测结果与蛋白酶敏感性实验高度一致。而Lewis血型抗原合成酶FUT3的G170S变异(△△G=9.8 kcal/mol)虽远离催化中心,仍通过破坏整体结构导致功能丧失,凸显了全局稳定性分析的重要性。

当前方法对固有无序区域(IDR)和翻译后修饰(如糖基化)的预测仍存挑战。未来需结合分子动力学模拟和化学伴侣筛选策略,推动从变异解读到精准治疗的转化。正如POMK(Walker-Warburg综合征相关激酶)案例所示(图7),结构导向的分析正为罕见病诊断开辟新路径。

 生物通微信公众号
生物通微信公众号
知名企业招聘