
-

生物通官微
陪你抓住生命科技
跳动的脉搏
基于知识图谱增强的图自编码器框架KEGNI精准推断细胞类型特异性基因调控网络
【字体: 大 中 小 】 时间:2025年09月24日 来源:Genome Biology 9.4
编辑推荐:
本研究针对单细胞RNA测序(scRNA-seq)数据推断基因调控网络(GRN)时存在高假阳性、缺乏因果性及细胞类型特异性信息整合不足的问题,开发了知识图谱增强框架KEGNI。该框架融合图自编码器(MAE)与知识图谱嵌入(KGE)模型,通过自监督学习和对比学习策略整合先验生物学知识。实验表明,KEGNI在BEELINE基准测试中优于8种现有方法,在配对多组学数据对比中与LINGER性能相当,可准确识别驱动基因并揭示细胞上下文特异性调控机制,为GRN推断提供了可扩展且高效的解决方案。
基因调控网络(GRN)揭示了细胞中基因与调控因子间复杂的相互作用关系,是理解生理和病理过程中细胞机制控制与动态变化的核心。近年来,单细胞RNA测序(scRNA-seq)技术的快速发展使得细胞类型特异性GRN的推断成为可能,但传统计算方法(如PIDC、GENIE3等)基于基因共表达模式进行推断,存在高假阳性问题,因为并非所有预测的相关性都代表因果关系。尽管深度学习模型(如scGeneRAI、STGRNS等)在捕获非线性依赖关系方面表现出优势,但如何有效整合外部知识并构建细胞类型特异性的GRN仍是一个挑战。
在此背景下,上海交通大学的研究团队在《Genome Biology》发表了题为“KEGNI: knowledge graph enhanced framework for gene regulatory network inference”的研究论文,提出了一种知识图谱增强的GRN推断框架KEGNI。该框架通过整合图自编码器和知识图谱嵌入技术,有效提升了GRN推断的准确性和生物学可解释性。
研究采用的关键技术方法包括:基于k近邻(k-NN)算法从scRNA-seq表达数据构建基础GRN;使用掩码图自编码器(MAE)通过自监督学习重构被掩码基因的表达特征以学习基因表征;从KEGG PATHWAY数据库和CellMarker 2.0提取细胞类型特异性标记基因构建知识图谱,并采用ComplEx模型进行知识图谱嵌入;通过多任务学习联合优化MAE和KGE目标函数。所用数据来源于公共数据库和已发表研究,包括BEELINE基准数据集、GEO收录的微胶质细胞和胰腺胰岛数据集。
KEGNI由两个核心组件构成:掩码图自编码器(MAE)模型和知识图谱嵌入(KGE)模型。MAE模型从scRNA-seq数据中构建基础GRN,通过随机掩码节点特征并重构其表达水平以学习基因间关系;KGE模型则整合KEGG通路和细胞类型特异性标记基因构建的知识图谱,采用对比学习和负采样策略进行嵌入学习。通过多任务学习将两类模型的损失函数结合,共同优化共享基因的表征,最终输出量化调控互作强度的权重矩阵。

研究使用BEELINE框架评估KEGNI性能,涵盖5个小鼠和2个人类细胞系的7个scRNA-seq数据集,以早期精确率(EPR)为主要指标。结果显示,KEGNI在12项基准测试中表现最佳,其归一化EPR值显著高于PIDC、GENIE3、GRNBoost2等8种方法。独立运行的MAE模型也优于多数现有方法,表明自监督学习策略有效捕获了基因关系。此外,在利用配对scRNA-seq和scATAC-seq数据的PBMC数据集上,KEGNI与LINGER性能相当(AUROC分别为0.699和0.714),显著优于仅使用scRNA-seq的方法。
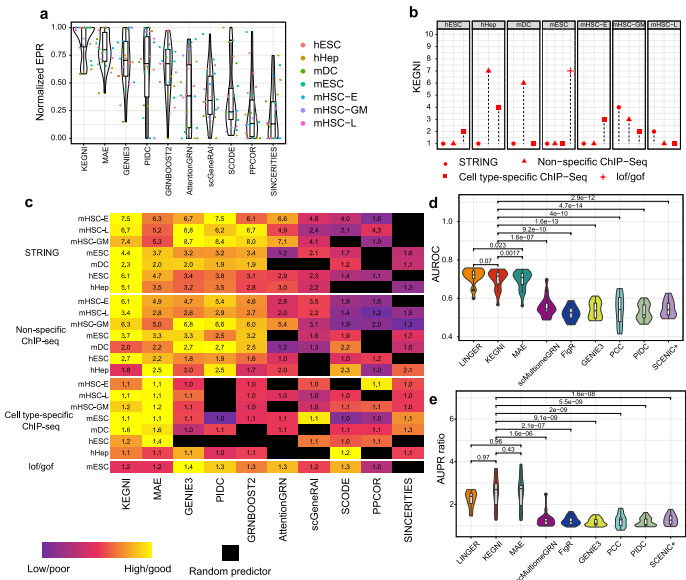
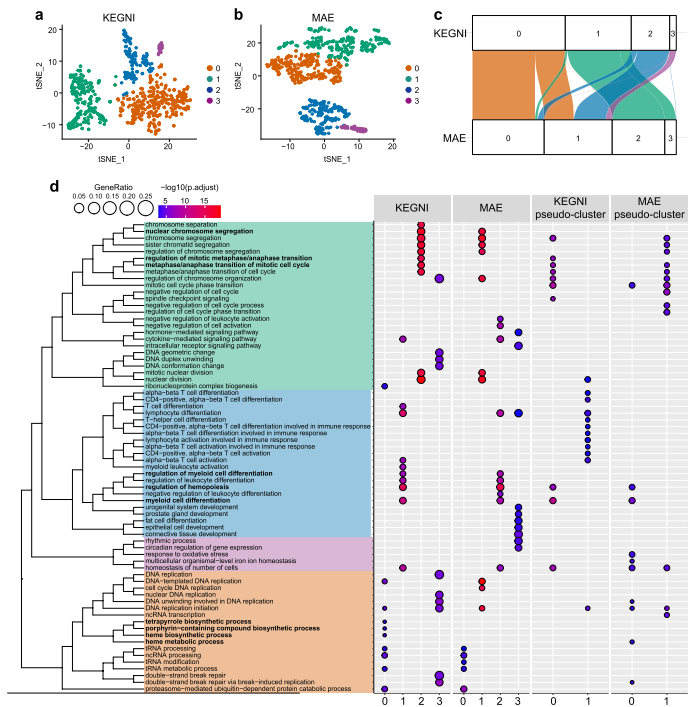
以小鼠造血干细胞红系谱系(mHSC-E)数据集为例,对KEGNI和MAE生成的基因嵌入进行聚类和GO富集分析。结果显示,KEGNI聚类结果更符合生物学背景,调整兰德指数(ARI)为0.575(MAE为0.407)。簇KEGNI_0显著富集“四吡咯生物合成过程”等术语,KEGNI_1富集“髓系细胞分化调控”等术语,与红细胞发育的关键过程一致,而随机伪聚类则无此特征,表明KEGNI捕获了具有生物学意义的模式。
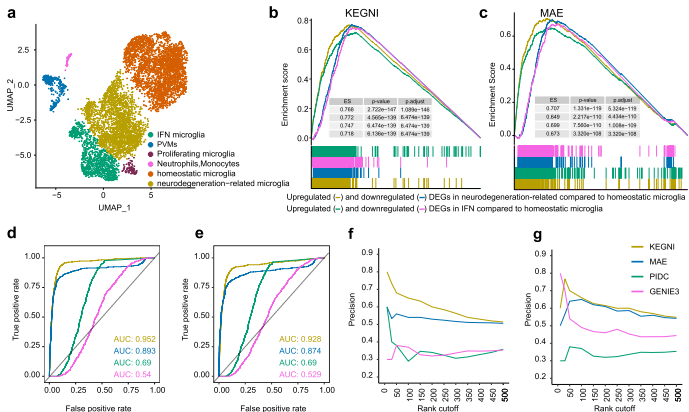
利用Cop1敲除微胶质细胞的scRNA-seq和ChIP-seq数据,以差异表达基因(DEG)和ChIP-seq靶基因为金标准,评估KEGNI对Cop1和Cebpb调控基因的预测能力。基因集富集分析(GSEA)显示,KEGNI的富集分数(ES)显著高于其他方法(调整p<0.05),AUROC达0.952和0.928。以ChIP-seq验证时,KEGNI在top-k精确率曲线中表现最佳,证实其能高置信度预测直接靶点。
在高脂饮食喂养小鼠的胰腺β细胞数据中,KEGNI通过调控评分识别Cd81low和Cd81high亚群的驱动基因。GSEA显示,Cd81low富集于胰岛素分泌调控相关通路,Cd81high富集于内质网应激反应通路。关键驱动转录因子如Mafa、Hmgb2、Jun和Neurod1在亚群中呈现差异调控网络:Mafa在Cd81high中调控炎症因子Bmp2;Hmgb2与炎症相关基因Mastl互作;Neurod1与Stat2和Id2的调控关系经ChIP-seq验证证实,揭示了β细胞在应激状态下的特异性调控机制。

KEGNI通过自监督学习与知识图谱引导的训练策略,有效整合scRNA-seq数据与先验知识,实现了细胞类型特异性GRN的精准推断。其在BEELINE基准测试中的优异表现证明了框架的可靠性,即使不依赖表观遗传数据也可达到与多组学方法相当的性能。局限性包括对细胞标记数据库的依赖、缺乏直接调控关系与因果性分析,以及未整合时间序列数据。未来可通过顺式调控元件分析或动态网络建模进一步优化。
作为一种通用的自监督学习框架,KEGNI为揭示复杂调控事件和基因表达变化提供了有力工具,有望在发育生物学和疾病机制研究中发挥重要作用。
 生物通微信公众号
生物通微信公众号
知名企业招聘