
-

生物通官微
陪你抓住生命科技
跳动的脉搏
GeneRAIN:基于深度学习的基因表达网络多维度表征及其在长链非编码RNA功能预测中的突破性应用
【字体: 大 中 小 】 时间:2025年09月24日 来源:Genome Biology 9.4
编辑推荐:
本研究针对当前基因表达深度学习模型在生物信息捕获广度与标准化方法上的局限性,开发了基于Transformer架构的GeneRAIN模型系列。通过创新性"Binning-By-Gene"标准化技术,利用410K人类bulk RNA-seq样本学习基因间表达关系,成功构建了超越传统方法的基因多维表征向量GeneRAIN-vec。研究证实该模型能有效捕获蛋白结构域、转录因子靶点、疾病关联等多样化生物属性,并实现了62.5 million个长链非编码RNA生物学属性的精准预测,为基因功能研究提供了全新范式。
在当今基因组学时代,理解基因功能及其与表型的复杂关系已成为生命科学研究的核心挑战。基因间的相互作用和调控过程构成了一张精密的网络,而破译这些关系对于解决生物学基本问题和推动生物医学应用——包括新疗法开发——具有关键意义。尽管深度学习技术尤其是Transformer模型和自监督学习的出现已经彻底改变了多个领域,但在基因表达数据分析中,我们对其学习生物信息的深度和广度仍知之甚少。
现有研究存在几个明显空白:大多数模型基于单细胞RNA-seq(scRNA-seq)数据训练,虽然能捕捉细胞异质性,却受限于高dropout率和技术噪音;相反,bulk RNA-seq虽然代表细胞混合物,但提供了更高的覆盖度和更精确的基因表达量化,且能捕获更广泛的表型谱和样本多样性,可能实现更高效的模型训练。然而,基于bulk RNA-seq的模型及其性能尚未得到充分探索。此外,虽然BERT模型通过随机掩码基因身份进行重建的方法被广泛使用,但其他架构如GPT模型在基因网络建模中的有效性仍未得到系统评估。
针对这些挑战,研究人员在《Genome Biology》上发表了题为"GeneRAIN: multifaceted representation of genes via deep learning of gene expression networks"的重要研究。该研究开发了GeneRAIN——一套基于Transformer的模型,通过410K人类bulk RNA-seq样本学习基因表达关系,采用创新的"Binning-By-Gene"标准化技术,捕获了表达之外的多样化生物信息。
关键技术方法
研究团队从ARCHS4数据库获取722,425个人类bulk RNA-seq样本,经过质控筛选出410,850个高质量样本。采用新型"Binning-By-Gene"标准化方法,将每个基因的表达值按样本间排名分配到2000个bin中,确保每个基因在模型输入中占据任何位置的概率相等。基于此建立了三种模型架构:BERT-Pred-Genes(预测被掩码基因身份)、BERT-Pred-Expr(预测被掩码的表达值)和GPT(基于高表达基因进行下一基因预测),统称为GeneRAIN(GR)模型。
模型架构与性能评估

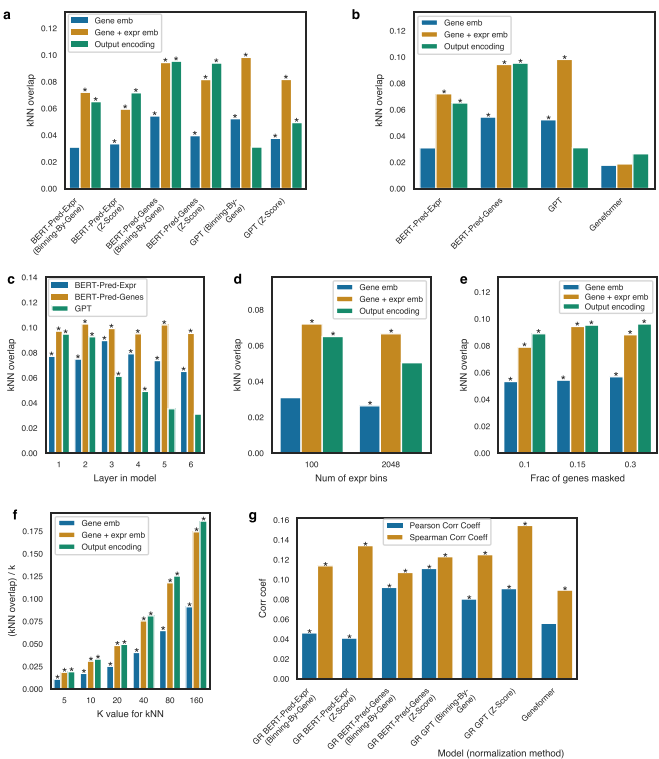
研究人员开发了Attribute Learning Index来综合评估模型学习基因生物属性的能力,该指数是调整兰德指数(ARI)、Fowlkes-Mallows指数(FMI)和标准化互信息(NMI)的平均值。结果显示,采用"Binning-By-Gene"标准化的模型在学习基因生物属性方面显著优于基于z-score的方法。
基因属性学习的多维表征
研究发现基因嵌入(gene embeddings)能够捕获丰富的生物信息。通过分析嵌入维度与不同基因属性的关联性,发现虽然不同基因属性在某些维度上有重叠,但总体上各属性关联的维度非常多样化。这与Geneformer模型形成鲜明对比,后者显示的多样性要少得多。
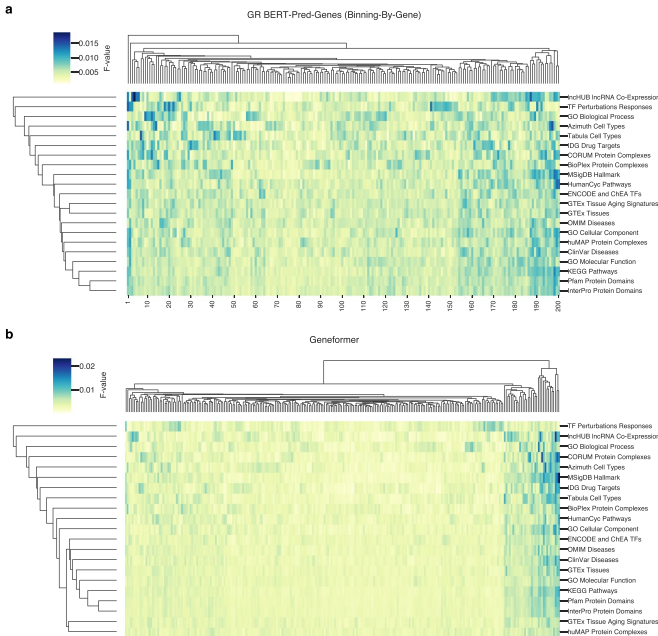
扰动响应学习能力评估
研究还评估了模型学习基因扰动后转录组响应能力。利用Replogle等人的大规模Perturb-seq数据集,研究发现所有GeneRAIN模型都包含了显著的转录组响应信息。采用"Binning-By-Gene"标准化的模型在捕捉扰动响应信息方面表现更优。
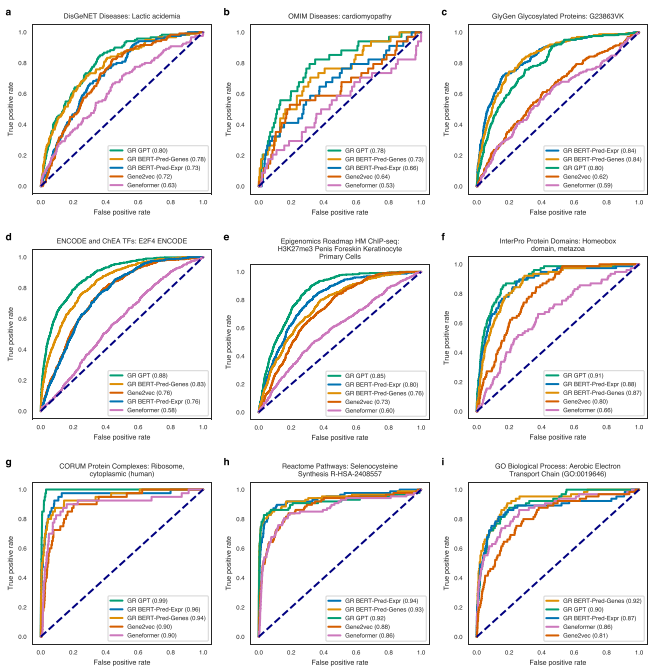
长链非编码RNA生物属性预测
研究最重要的应用之一是使用训练好的分类器预测长链非编码RNA(lncRNA)的生物属性。通过将lncRNA纳入训练数据,使其与编码基因共享相同的嵌入空间,研究人员使用主要在蛋白质编码基因上训练的分类器,成功对13,030个lncRNA做出了62.5 million个生物属性预测。
研究发现,基于蛋白质编码基因训练的分类器能够有效转移到lncRNA上,在疾病关联预测、转录因子靶点预测和基因本体(GO)预测等方面都表现出良好性能。这为理解lncRNA的生物学功能提供了强大工具。
研究结论与意义
GeneRAIN研究通过开发创新的标准化方法和多种Transformer架构,显著提升了从bulk RNA-seq数据中学习复杂基因关系的能力。研究表明,尽管模型仅基于基因表达数据训练,却能捕获广泛的生物信息,超越了单纯的表达关系。
该研究的核心贡献在于:首先,引入了"Binning-By-Gene"标准化方法,有效减少了基因表达分布差异带来的偏差;其次,证明了GPT架构在基因网络建模中的优越性;最后,展示了如何将基于蛋白质编码基因的知识有效转移到lncRNA上,为这一重要但功能特征较少的基因类别提供了大规模功能预测。
这些发现不仅深化了我们对Transformer和自监督学习在大型表达数据中应用的理解,也为生物学探索和发现开辟了新途径。未来研究可专注于整合其他数据类型、进一步优化模型架构,以及扩展这些模型的应用以解决更复杂的生物学问题。
研究的实际意义在于提供了一种全新的基因功能注释方法,特别是在lncRNA功能预测方面具有重要价值。通过GeneRAIN-vec这一多维基因表征,研究人员能够在无需先验知识的情况下,对各种基因属性进行准确预测,为功能基因组学研究提供了强大工具。

 生物通微信公众号
生物通微信公众号
知名企业招聘